Introduction
Materials and Methods
채집 지역 선정 및 샘플 채집
내생미생물 분리
내생미생물 동정
DNA 추출과 metagenomic 분석
Network analysis
Results and Discussion
배양성 내생미생물
난배양성 내생미생물
Diversity analysis
Network analysis
Conclusion
Introduction
천궁(Cnidium officinale)은 다년생 산형과에 속하는 식물로 보혈, 진정, 진통의 효과가 있는 것으로 알려져 있다(Kim et al., 2020). 실제로 천궁은 항산화 능력이 뛰어나 기능성 식품이나 화장품 원료로 이용되고 있다(Oh et al., 2010). 천궁의 구근 추출물은 팔카린디올(falcarindiol), 리구스티라이드(ligustilide) 등의 항염증 및 항암에 효과가 있는 성분을 포함하고 있다는 연구가 보고되었다(Bae et al., 2011). 훼룰린산(ferulic acid)은 활성산소를 중화하는 항산화, 항혈전증 등의 기능을 한다(Ou and Kwok, 2004). 이 외에도 혈압 조절이나 비타민 결핍, 월경 장애 등에도 탁월한 효과가 있는 것으로 알려져 있다(Jeong et al., 2009).
천궁은 중국이 원산지로 한국, 중국, 일본의 일교차가 큰 해발 400 m 내외의 준고랭지가 재배 적지이다(Jang et al., 2019). 하지만 지난 100년 동안 우리나라의 평균기온은 1.5℃ 상승하였으며 IPCC (기후변화 관련 정부간 협의체)는 현재 추세로 온실가스를 배출한다면 2100년에는 평균기온이 5.7℃가 상승할 것으로 예측하였다(Seo et al., 2016). 기온상승이 지속된다면 천궁재배가능지역도 지속적으로 감소되며 전국적으로 분포되어 있던 천궁의 재배지는 2090년대가 되면 일부 강원도 산간지역에서만 재배가 가능할 것으로 예측된다(Kim et al., 2015). 한국에서는 2021년 기준 재배면적의 90%가 경상북도에서 재배되고 있으며 이 중 88%가 영양군에서 재배되고 있다(KOSTAT, 2021a, 2021b). 따라서 천궁의 생산지역은 좁은 지역에 집중되어 있기 때문에 기온 상승으로 인해 천궁 재배가능 지역이 변화하면 천궁 생산량이 급격히 감소할 것으로 사료되며 이에 대한 생산량 증진 대책이 필요하다.
식물은 근권에 있는 다양한 미생물과 상호작용을 하며 양분흡수를 원활히 하거나 각종 환경스트레스에 대한 저항능력을 증진시킬 수 있다. 이러한 미생물을 식물 내생 미생물이라고 한다. 내생미생물의 대표적인 작용으로는 indole-3-acetic acid (IAA)와 같은 식물 생육 촉진 호르몬을 분비하거나 토양내 불용성 질소, 인, 철 등을 식물이 사용가능한 형태로 전환시키는 작용을 하기도 하며 병원성 곰팡이와 길항작용을 하는 등의 활동을 통해 식물의 생육을 증진시킬 수 있다(Lodewyckx et al., 2002; Lee et al., 2023). 또한, 유해물질을 분해하는 미생물을 포플러 시험지에 접종하였을 때 환경유해성 물질을 분해하여 식물의 생육 증진 및 환경정화를 하는 등 다양한 기능을 가지는 유용미생물 발굴에 대한 폭넓은 연구가 진행되고 있다(Majeed et al., 2015; Doty et al., 2017).
미생물은 식물과의 상호작용 외에도 미생물 사이의 상호작용도 복잡하게 연결되어 있으며 식물의 생육환경 및 생태계 유지에 필요한 다양한 대사작용이 이루어진다(Baldrian, 2017). 이렇게 관련성이 깊은 미생물 군을 module (또는 cluster)라고 하며 근권에는 여러 개의 module이 존재한다. 특정 미생물은 여러 개의 module과 상호작용을 하기도 하고 많은 종의 미생물과 깊은 연관성이 있을 수 있다. 이러한 미생물을 keystone taxa라고 하며 토양 미생물 군집의 기능에 중요한 역할을 한다(Banerjee et al., 2018).
따라서 본 연구에서는 미생물 제제를 활용한 천궁 생산성 증진연구의 기초자료를 제공하기 위해 천궁 최대 생산지인 태백, 정선, 봉화, 제천 지역의 5개 농가에서 재배되고 있는 천궁의 내생미생물을 분리 및 동정하고 근권 미생물 차이와 기능적 특성을 분석하였다. 또한, 배양성 미생물 외에도 next generation sequencing (NGS)을 활용한 metagenomic 분석을 통해 천궁의 근권조직에 존재하는 난배양성 미생물 군집의 주요 특징을 파악하고 천궁 생장에 중요한 영향을 주는 keystone taxa를 분석하였다.
Materials and Methods
채집 지역 선정 및 샘플 채집
천궁의 고유 내생미생물을 분석하기 위해 다양한 지역과 해발고도를 고려하여 5개의 재배지를 선정하였다. 채집 지역은 강원도 정선, 강원도 태백1, 강원도 태백2, 충청북도 제천, 경상북도 봉화이며 지상부의 발달이 우수하고 괴경 형성이 뛰어난 우량 개체 천궁을 각각 3개체씩 채집하였다. 채집한 천궁은 흐르는 수돗물에 24시간동안 세척하여 표면의 흙을 제거하고 1% NaClO 3분, 70% 에탄올에 2분 동안 표면 살균을 진행한 후 멸균수로 세척하였다. 클린벤치 내에서 멸균한 petri dish와 filter paper를 사용하여 상온에서 표면건조를 진행하였다. 이 후 멸균 매스를 사용하여 표피를 제거하고 작게 파쇄하여 실험에 사용하였다.
내생미생물 분리
배양이 가능한 내생미생물 분리를 위해 천궁 조각을 멸균한 증류수에 넣고 26.5℃에서 72시간 동안 배양하였다. 200 µL의 배양액을 tryptic soy agar (TSA), nutrient agar (NA), Luria-Bertani agar (LA) 배지에 각각 평판도말하고 39℃에서 3일간 배양하였다. 배양된 균주들은 형태학적으로 다른 균주를 선택하여 순수 분리가 될 때까지 획선도말을 진행하였다. 분리된 균주는 추후 연구를 위해 50% glycerol 용액에 1 : 1로 혼합하여 -20℃에서 보관하였다.
내생미생물 동정
미생물 동정은 ChunLab, Inc. (Korea)에 의뢰하여 진행하였다. 미생물에서 DNA를 추출한 후 universal primer 27F (5’-AGAGTTTGATCMTGGCTCAG-3’)와 universal primer 1492R (5’-TACGGYTACCTTGTTACGACTT-3’)을 사용하여 polymerase chain reaction (PCR)을 진행하였다. 각 단계는 초기 변성(initial denaturation, 95℃, 5 min)을 1회를 하고, 변성(denaturation, 95℃, 0.5 min), 결합(annealing, 55℃, 2 min), 증폭(extension, 68℃, 1.5 min)을 30회 반복한 후, 최종 증폭(final extension, 68℃, 10 min)을 1회 진행하였다. PCR 증폭 산물의 반응하지 않은 PCR primer와 deoxyribonucleotide triphosphates (dNTPs)는 Montage PCR clean up kit (Merck Millipore, USA)를 사용하여 제거하였으며 최종적으로 785F (5’-GGATTAGATACCCTGGTA-3’)과 907R (5’-CCGTCAATTCMTTTRAGTTT-3’) primer를 사용하여 시퀀싱하였다(Manoharan et al., 2020). 시퀀싱에 사용한 kit와 장비는 Big Dye Terminator v3.1 cycle sequencing kit (Applied Biosystems, USA)와 3730XL automated DNA sequencing system (Applied Biosystems, USA)을 사용하였다. 시퀀싱 데이터는 BLASTn tool로 NCBI의 standard databases와 대조하여 동정을 진행하였다(NCBI, 1988).
DNA 추출과 metagenomic 분석
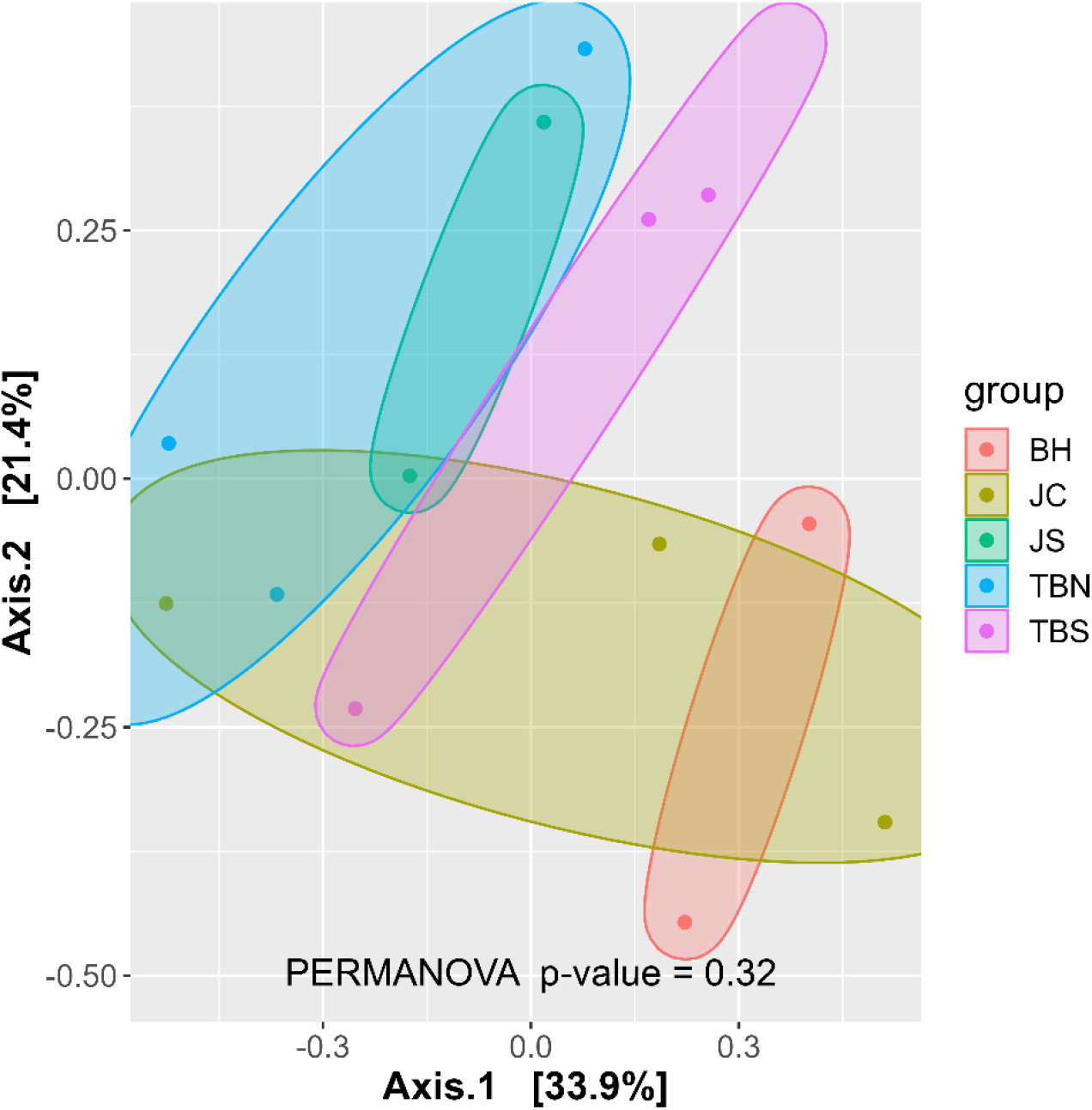
실험실에서 배양하기 어려운 난배양성 미생물의 군집구조를 확인하기 위해 metagenomic 분석을 수행하였다. 표면소독이 끝난 천궁은 DNA는 Maxwell® RSC PureFood GMO and Authentication kit (Promega, Spain)를 사용하여 추출하였다. 추출한 DNA는 V3 - V4영역을 PCR한 후 CleanPCR (CleanNA)로 정제하였다(Wu et al., 2016). 샘플의 퀄리티는 DNA 7500 chip을 사용하여 bioanalyzer (Agilent, USA)로 확인하였다. PCR이 끝난 샘플은 ChunLab, Inc. (Korea)에 의뢰하여 Illumina MiSeq system (Illumina, USA)을 사용하여 sequencing하였다. Sequencing된 rawdata는 QIIME2의 DADA2를 사용하여 low-quality sequences를 제거하고 amplicon sequence variants (ASVs)를 얻었다(Callahan et al., 2016; Bolyen et al., 2019). ASVs는 SILVA (ver. 138)을 사용하여 동정을 진행하였으며 계통수는 SATé-enabled phylogenetic placement (SEPP)와 SILVA (ver. 128)을 사용하여 분석하였다(Janssen et al., 2018; Robeson et al., 2021). Beta diversity 분석은 weighted UniFrac matrix를 사용하여 principal coordinates analysis (PCoA)로 시각화 하였으며 통계분석을 위해 permutational multivariate analysis of variance (PERMANOVA) 분석을 수행하였다(Anderson, 2001; Hamady et al., 2010; Halko et al., 2011).
Network analysis
분석된 taxa들의 상호작용을 분석하기 위한 network 구축과 network의 특징을 분석은 molecular ecological network analysis pipeline (MENA) (http://ieg4.rccc.ou.edu/mena accessed on 20 December 2022)을 사용하여 분석을 진행하였다(Deng et al., 2012). Cutoff threshold value는 0.31로 하였으며 inference of direct and indirect relationships with effective copula-based transitivity (iDIRECT)를 사용하여 간접적 영향에 의한 network를 제거하였다(Xiao et al., 2022). Network 구축 방법은 Pearson correlation과 leading eigenvector of the community matrix를 사용하였으며 Cytoscape (ver. 3.9.1)를 사용하여 시각화하였다(Shannon et al., 2003; Newman, 2006). Network 분석을 통해 얻은 Zi와 Pi 값은 각각 2.5와 0.62를 기준으로 중요도를 평가하였다. Module hubs (Zi > 2.5, Pi < 0.62)는 자신의 모듈 내에서 많은 연결을 보이는 종이다. Connectors (Zi < 2.5, Pi > 0.62)는 다른 모듈과의 많은 연결을 보이는 taxa를 의미한다. Network hubs (Zi > 2.5, Pi > 0.62)는 module hubs와 connector의 특징을 모두 가진 taxa를 의미한다. 또한, phylogenetic investigation of communities by reconstruction of unobserved states 2 (PICRUSt2)와 Kyoto encyclopedia of genes and genomes (KEGG) database 사용하여 각 샘플의 박테리아 기능 예측 분석을 수행하고 각 module과 Mantel test를 통해 module의 기능을 분석하였다(Douglas et al., 2020; Kanehisa et al., 2022).
Results and Discussion
배양성 내생미생물
배양의존적 방법으로 내생미생물을 분리한 결과, 강원도 정선에서 8균주, 강원도 태백1에서 12균주, 강원도 태백2에서 1균주, 충청북도 제천에서 4균주를 분리하였으며 총 25균주를 분리하였다. 경상북도 봉화에서는 내생균이 분리되지 않았다. 분리된 균주의 16S rRNA 분석을 통한 동정 결과 총 8종이 확인되었다(Table 1).
Table 1.
Results of identification for each isolated strain from Cnidium officinale Makino.
| Species | GeneBank No.z | Jeongseon | Jecheon | Teabaek | Total |
| Enterobacter ludwigii | NR_042349 | 3 | 4 | 0 | 7 |
| Pantoea agglomerans | NR_114735 | 2 | 0 | 8 | 10 |
| Paenibacillus provencensis | NR_044179 | 2 | 0 | 0 | 2 |
| Strenotrophomonas pavanii | NR_118008 | 0 | 0 | 2 | 2 |
| Bacillus firmus | NR_112635 | 0 | 0 | 1 | 1 |
| Bacillus aerophilus | NR_042339 | 1 | 0 | 0 | 1 |
| Bacillus velezensis | NR_075005 | 0 | 0 | 1 | 1 |
| Bacillus wiedmannii | NR_152692 | 0 | 0 | 1 | 1 |
동정결과, Enterobacter ludwigii와 Pantoea agglomerans가 배양이 가능한 주요 내생미생물인 것으로 확인되었다. 실제로 E. ludwigii는 선행연구에서 천궁의 생육을 촉진시키는 IAA를 분비하는 것으로 확인되었으며 천궁에 접종을 하였을 때 생중량을 증가시키는 효과가 있었다(Jeon et al., 2023). 또한 P. agglomerans도 IAA와 siderophore 분비, 인산가용화능력 등과 같은 식물 생육증진과 관련된 DNA를 가지는 것으로 보고되었으며 선행 연구에서도 천궁에서 분리된 P. agglomerans가 병원성 곰팡이에 대한 길항능 및 식물생육증진에 효과가 있는 protease와 cellulase를 분비하는 것을 확인하였다(Shariati et al., 2017; Seo, 2022). 하지만 P. agglomerans는 아직까지 실제 천궁의 생육에 주는 영향과 관련된 연구가 없어 후속연구가 필요한 것으로 판단된다.
난배양성 내생미생물
Illumina Miseq system을 통해 sequencing한 rowdata를 forward sequence는 240 bp, reverse sequence는 220 bp에서 trim을 진행하여 평균 46,579 ± 4,212 reads를 획득하였다. Trim을 진행한 데이터는 PCR과정에서 문제가 발생한 샘플 2개와 chloroplast, mitochondria DNA를 filtering 하여 최종적으로 13개의 샘플에서 평균 462 ± 275 reads를 분석에 사용하였다.
문 수준에서는 모든 샘플에서 12개의 문을 확인할 수 있었으며 Proteobacteria가 평균 46.3 ± 9.6%로 가장 우점하였으며, Actinobacteriota가 평균 35.1 ± 10.1%로 두 번째로 우점하는 것으로 분석되었다. 목 수준의 미생물 군집 분석에서는 총 48개의 목을 확인할 수 있었다. 분석결과 5개의 지역에서 모두 확인된 목은 9개이며 이 중 5개의 목은 90% 이상의 샘플에서 확인할 수 있었다. 5개의 목은 Rhizobiales가 32.8 ± 11.6%로 가장 우점하였으며 Streptomycetales가 14.8 ± 11.9%로 두 번째로 우점하였고, Saccharimonadales, Micromonosporales, Sphingomonadales 순서로 우점하는 형태를 보였다(Fig. 1). 따라서 위의 5개의 문이 천궁이 고유하게 형성하는 주요 미생물 군집인 것으로 판단된다.
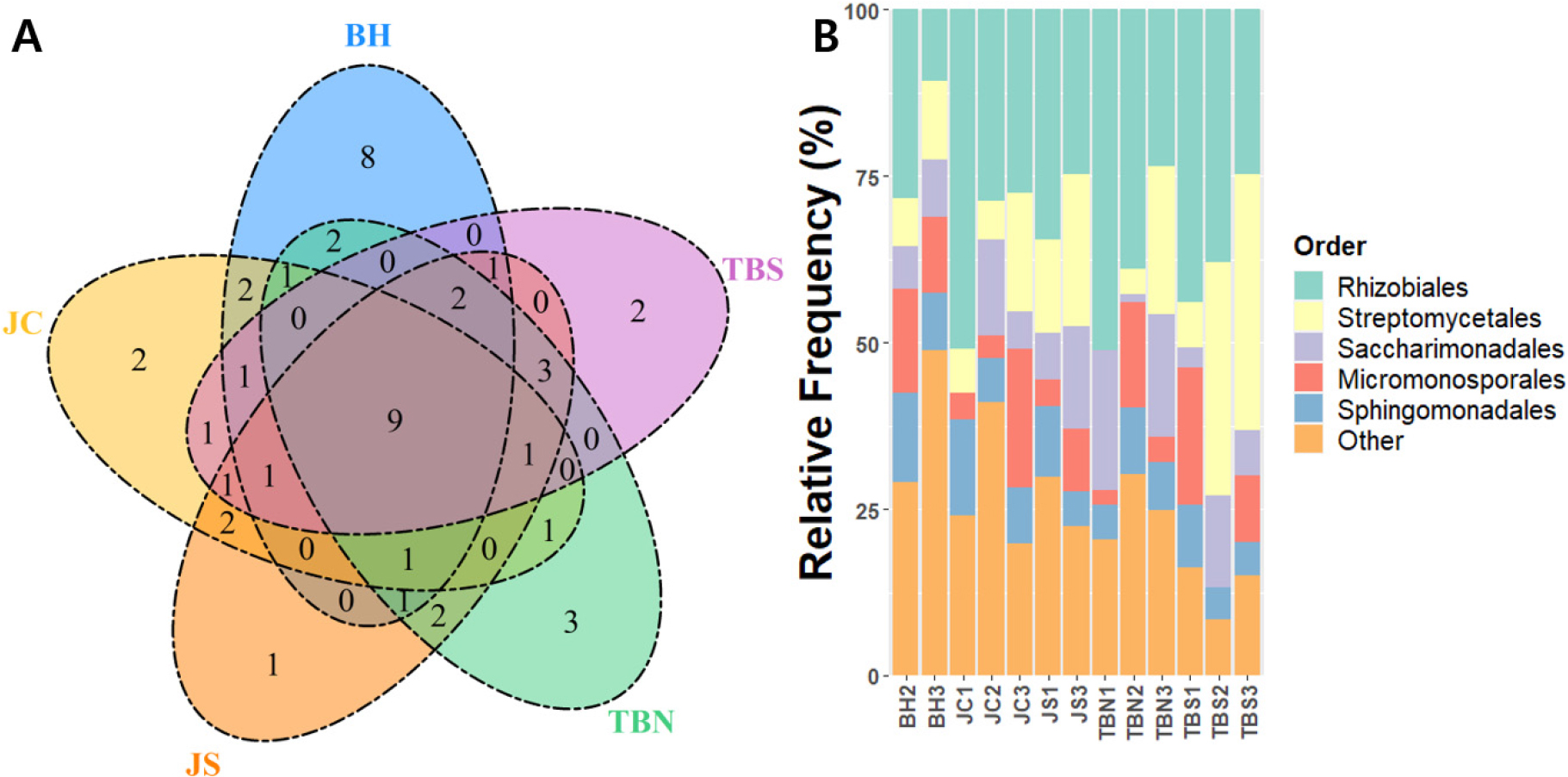
Fig. 1.
Metagenomic analysis result at order level. (A) Venn diagram showing the number of shared and unique operational taxonomic units (OTUs) for each site. (B) Bar plot showing the relative frequency of each OTUs at order level. The low frequency OTUs (mean frequency < 7%) were grouped into the “Other” group. BH, Bonghwa; TBN, Taebaek 1; TBS, Taebaek 2; JS, Jeongseon; JC, Jecheon.
가장 우점하는 목으로 나타난 Rhizobiales 목은 질소고정능력과 관련이 있는 것으로 알려져 있으며 주로 콩과식물의 뿌리에서 공생하는 것으로 알려져 있다(Carvalho et al., 2010). 하지만 대부분의 Rhizobiales 목은 콩과식물이 아닌 식물과도 상호작용을 하는 능력이 있고 기주에게 영향을 준다(Garrido-Oter et al., 2018). 따라서 천궁의 생산량과도 깊은 관련이 있을 것으로 사료된다. Streptomycetales 목은 분석결과 모두 Streptomyces 속으로 분류되었다. Streptomyces 속은 토양 속에서 생활사를 이어가다가 식물의 근권에서 분비하는 mucilage 등에 의해 식물과 상호작용을 한다. 이 중 일부는 식물의 조직 내로 침투하여 영양분을 식물에게 전달하거나 생육 촉진 호르몬 분비 및 길항작용 등을 통해 식물의 생육을 증진시킨다(Sousa and Olivares, 2016; Amaresan et al., 2018). 이 외에 Saccharimonadales, Micromonosporales, Sphingomonadales 목들도 천궁의 뿌리조직 내에서 확인되었으며 내생미생물의 대사작용에 관여하는 것으로 판단된다.
Diversity analysis
각 지역 간의 천궁 내생미생물의 구조에서 차이가 있는지 통계적으로 분석하기 위해 beta diversity분석을 진행하였다. Beta diversity 분석 결과에서 PERMANOVA p-value 값이 0.32로 유의수준(p-value < 0.05)보다 커 beta diversity에서 차이가 없는 것으로 분석되었다. 또한 PCoA를 사용하여 시각화한 그래프에서도 지역 간의 특징을 보이지 않았다(Fig. 2). 따라서 각 지역의 천궁 내생미생물의 군집구조는 지역에 상관없이 유사한 구조를 보이는 것으로 판단된다.
Network analysis
Classified 된 taxa의 상호작용을 분석하기 위해 MENA를 사용하여 network analysis를 수행한 결과 총 38개의 node와 3개의 module을 확인할 수 있었다. 또한 상위 5개의 우점 목 또한 포함되는 것으로 분석되었다. 분석결과 module hubs와 network hubs로 보이는 taxa는 확인되지 않았다. 하지만 총 8개의 taxa가 connectors로 분류되었으며, Rhizobiales 목에서 3개, Streptomycetales 목에서 1개, Saccharimonadales 목에서 1개, 그리고 기타 다른 목에서 3개의 taxa가 keystone taxa인 것으로 확인되었다(Fig. 3). Connectors 사이에서의 우점도는 Streptomycetales 목이 14.8 ± 11.9%로 가장 우점하여 여러 모듈에 영향을 줄 것을 보이며 그 다음으로 Rhizobiales, Saccharimonadales 목이 우점하는 형태를 보였다(Table 2). 상위 3개분류군의 우점도는 20.6%를 차지하였다.
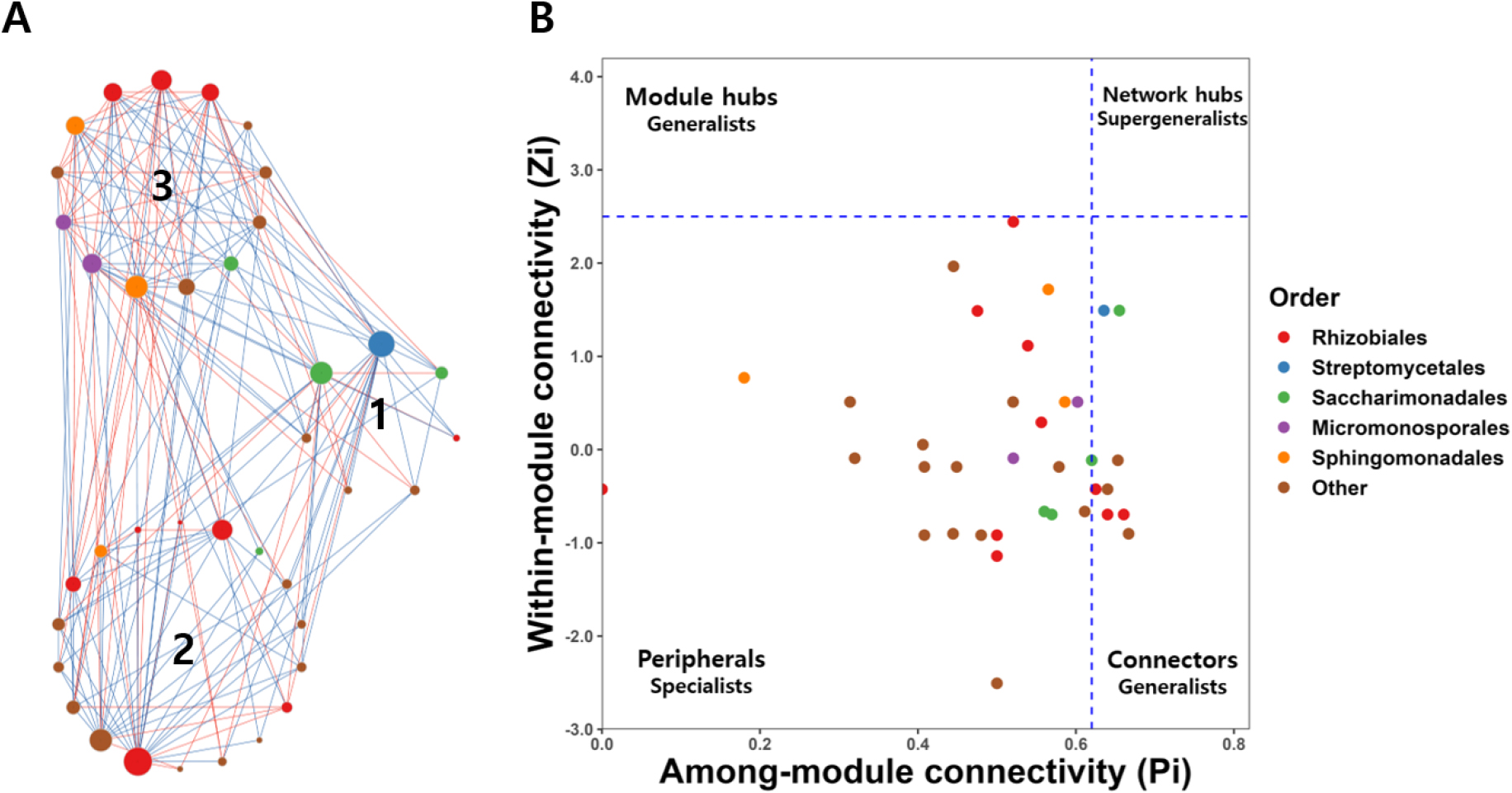
Fig. 3.
Result of network analysis. (A) Correlation of each operational taxonomic units (OTUs). Node size indicates degree of each OTUs (number of network with other nodes). Red line indicates negative correlation and blue line indicates positive correlation. Each number indicates each module. (B) Zi-Pi plot showing the distribution of nodes based on their topological roles in network. The threshold values of Zi and Pi for categorizing nodes are 2.5 and 0.62, respectively.
Table 2.
Information of each connector.
| Taxa | Module | Order | Family | Gene | %z |
| OTU3 | 1 | Streptomycetales | Streptomycetaceae | Streptomyces | 14.8 ± 11.9 |
| OTU7 | 3 | Rhizobiales | Xanthobacteraceae | Bradyrhizobium | 5 ± 6.3 |
| OTU16 | 1 | Saccharimonadales | LWQ8 | LWQ8 | 5.8 ± 5.5 |
| OTU18 | 2 | Caulobacterales | Caulobacteraceae | Caulobacter | 1.1 ± 1.8 |
| OTU21 | 3 | Rhizobiales | Rhizobiaceae | Mesorhizobium | 1.6 ± 1.9 |
| OTU72 | 1 | Tepidisphaerales | Tepidisphaeraceae | Tepidisphaera | 0.3 ± 0.6 |
| OTU102 | 2 | Rhizobiales | Xanthobacteraceae | - | 0.4 ± 1.2 |
| OTU176 | 2 | Azospirillales | Azospirillaceae | Azospirillum | 0.1 ± 0.3 |
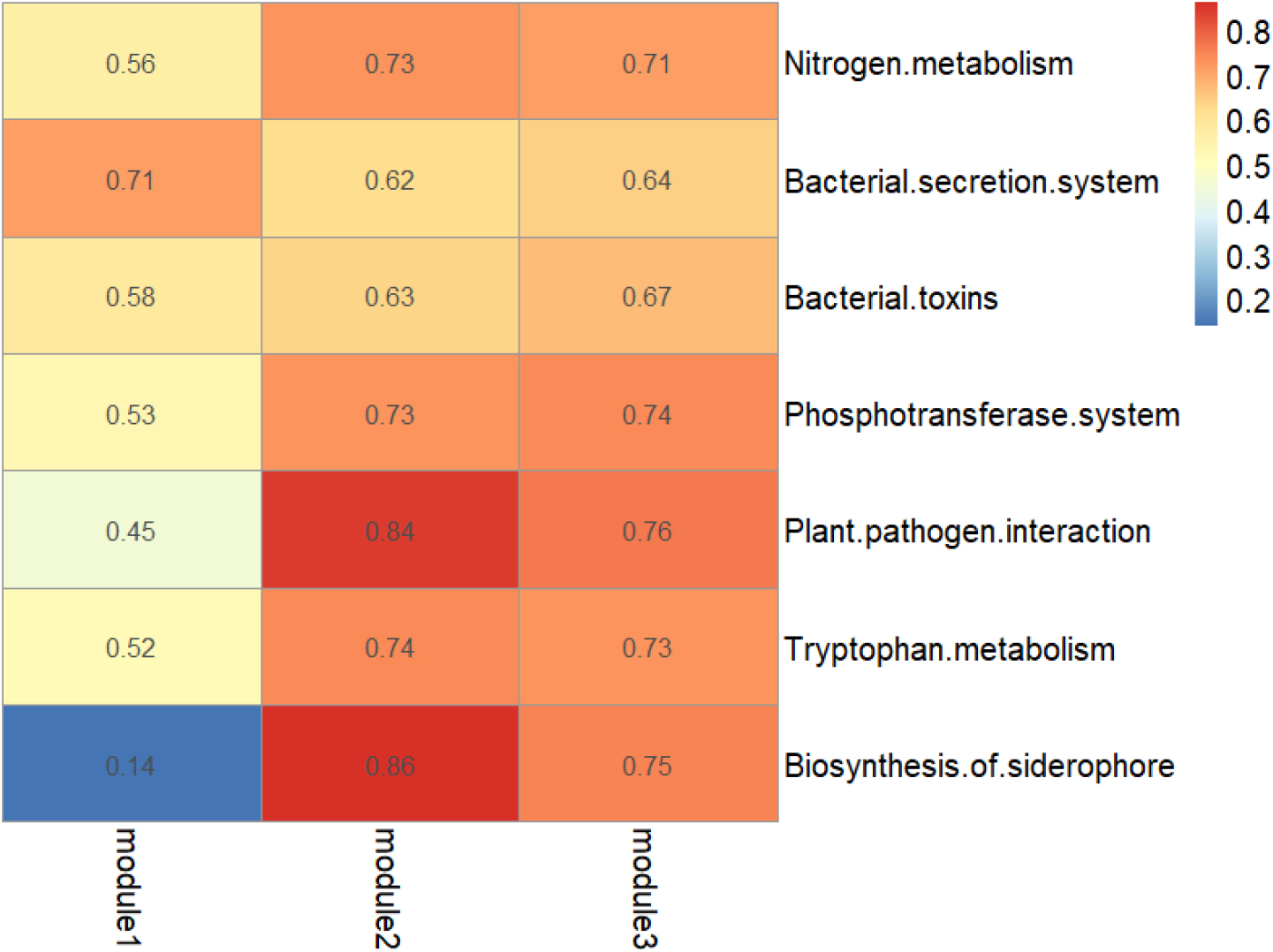
PICRUSt2로 각 샘플의 박테리아 기능 예측 분석을 수행하고 각 module과 Mantel test를 수행하여 module의 기능을 분석하였다. 대부분의 기능에서 양의 상관관계를 보였지만 module 1의 경우 bacterial secretion system이 다른 module에 비해 높은 상관관계를 보였다. 반면에 nitrogen metabolism, phosphotransferase system, plant-pathogen interaction, tryptophan metabolism, biosynthesis of siderophore 기능은 module 2와 module 3에서 module 1보다 높은 상관관계를 보였다. 특히 module 2의 경우 plant-pathogen interaction과 siderophore의 생합성 능력이 높은 상관관계를 보이는 것을 확인할 수 있었다(Fig. 4).
Nitrogen metabolism은 식물의 질소고정과 흡수에 관련이 있으며 bacterial secretion system은 미생물이 식물의 조직 내에 침투하여 내생미생물이 되는 것에 기여하며 실제로 효율적인 질소고정과 관련이 있다는 보고가 있다(Lucke et al., 2020; Mazumdar et al., 2020). 이 외에도 병원성 곰팡이에 대한 길항작용과도 관련이 있다. 또한 bacterial toxins와 plant-pathogen interaction도 길항작용과 관련이 있다(Dutta and Podile, 2010; Joshi et al., 2020). Phosphotransferase system은 당 기질의 흡수와 인산화 촉매 작용에 관여한다. 선행 연구에서 Phosphotransferase 단백질은 탄소 및 질소의 대사와 biofilm 형성을 조절한다고 보고되었다(Gao et al., 2017). siderophore의 생합성 능력은 철의 가용화와 길항능력과 관련이 있다(Saha et al., 2016; Gao et al., 2017). 마지막으로 tryptophan metabolism은 IAA의 전구물질 대사이며 IAA 생성을 조절하여 식물생육을 증진시킨다(Goswami et al., 2014).
Conclusion
본 연구에서는 기온상승으로 인해 천궁의 재배지역의 변화에 대응하기 위해 천궁의 배양성 및 난배양성 내생미생물을 분석하였다. 배양성 미생물들은 기능성 및 생리적 특성을 조사하고 분석한다면 미생물 제재로 활용가능성이 높으며 기후변화에 대응하여 천궁의 생육증진에 활용할 수 있다(Gupta et al., 2015). 또한, 천궁의 난배양성 내생미생물을 분석한 결과 모든 지역의 천궁 내생 미생물 군집의 beta diversity에서 유의한 차이가 없는 것으로 분석되었다. 따라서 천궁은 지역에 상관없이 고유한 내생 미생물 군집을 형성하는 것으로 판단된다. 고유 미생물 기능을 분석에서는 식물 생육 증진에 관련된 특성들은 각 천궁 내생미생물 module과 양의 상관관계가 있는 것으로 분석되었으며 재배지역에서 keystone taxa의 감소 및 부재에 의해 module의 형성이 부진할 경우 천궁의 생산량의 감소로 이어질 수 있다. 이에 대응하기 위한 유용미생물의 인공접종 등의 방법과 지속적인 모니터링을 고려할 필요가 있을 것으로 사료된다.